Patterning of the ascidian neural tube
Publications from this project:
- Hudson C, Yasuo H. (2021). Neuromesodermal Lineage Contribution to CNS Development in Invertebrate and Vertebrate Chordates. Genes doi: 10.3390/genes12040592. hal-03215448v1.
- Hudson C, Esposito R, Palladino A, Staiano L, Ferrier D, Faure E, Lemaire P, Yasuo H and Spagnuolo A. (2019). Transcriptional regulation of the Ciona Gsx gene in the neural plate. Dev Biol. doi: 10.1016/j.ydbio.2018.12.013. hal-02115329v1.
- Hudson C (2019). A simple method to identify ascidian brain lineage cells at neural plate stages following in situ hybridisation. Methods in brain development. 2nd Methods in Molecular Biology series, Springer Nature. Methods Mol Biol., 2047:325-345. doi: 10.1007/978-1-4939-9732-9_18. hal-02322828v1
- Esposito R#, Yasuo H#, Sirour C, Palladino A, Spagnuolo A and Hudson C#. (2017). Patterning of brain precursors in ascidian embryos. Development 144:258-264. DOI: 10.1242/dev.142307. hal-01430785v2.
- Hudson C. (2016). The central nervous system of ascidian larvae. Wiley Interdisciplinary Reviews: Developmental Biology. doi: 10.1002/wdev.239. hal-01336969v1
- Hudson C, Sirour C and Yasuo H. (2015). Snail mediates medial-lateral patterning of the ascidian neural plate. Biol., 403, 172-179. DOI: 10.1016/j.ydbio.2015.04.026. hal-01155499v1.
- Haupaix N, Abitua PB, Sirour C, Yasuo H, Levine M and Hudson C. (2014). Ephrin-mediated restriction of ERK1/2 activity delimits the number of pigment cells in the Ciona Dev. Biol. 394:170-80. DOI: 10.1016/j.ydbio.2014.07.010. hal-02115480v1.
- Nishitsuji K, Horie T, Ichinose A, Sasakura Y, Yasuo H and Kusakabe TG. (2016). Cell lineage and cis-regulation for a unique GABAergic/glycinergic neuron type in the larval nerve cord of the ascidian Ciona intestinalis. Growth Differ. 54:177-86. DOI : 10.1111/j.1440-169X.2011.01319.x. hal-04103275v1.
- Hudson C, Ba M, Rouvière C and Yasuo H. (2011). Divergent mechanisms specify chordate motoneurons: evidence from ascidians. Development 138: 1643-52. DOI: 10.1242/dev.055426. hal-02115498v1.
- Hudson C and Yasuo H. (2008). Similarity and diversity in mechanisms of muscle fate induction between ascidian species. Cell. 100:265-77. DOI: 10.1042/BC20070144. hal-02115560v1. Review.
- Hudson C, Lotito S and Yasuo H. (2007). Sequential and combinatorial inputs from Nodal, Delta2/Notch and FGF/MEK/ERK signalling pathways establish a grid-like organisation of distinct cell identities in the ascidian neural plate. Development 134: 3527-37. DOI: 10.1016/j.ydbio.2006.08.075. hal-02115570v1.
- Hudson C and Yasuo H. (2005). Patterning across the ascidian neural plate by lateral Nodal signalling sources. Development 132:1199-210. DOI: 10.1242/dev.01688. hal-02115579v1.
- Hudson C, Darras S, Caillol D, Yasuo H and Lemaire P. (2003). A conserved role for the MEK signalling pathway in neural tissue specification and posteriorisation in the invertebrate chordate, the ascidian Ciona intestinalis. Development 130:147-59. DOI: 10.1242/dev.00200. hal-02115682v1.
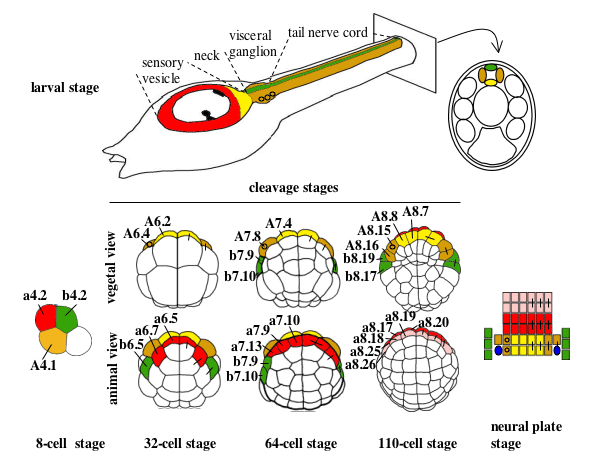
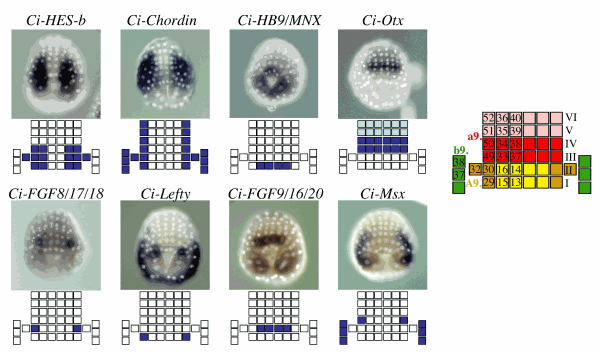
Figure 1. Cell lineages of the ascidian larval CNS (from Hudson and Yasuo, 2005). Cell lineages are indicated as follows: the a-line is coloured red (anterior sensory vesicle precursors) or pink (anterior epidermis and pharynx/neurohypothesis precursors); b-line is green and A-line yellow at the 8-cell stage and light yellow (medial cells) or tan (lateral cells) from the 32-cell stage. Bars connecting two blastomeres on the right-hand side of the drawings indicate sister cell relationship.Small circles in the lateral-A-line precursors on the left hand side indicate that these blastomeres give rise to the motoneurones; the final position of the motoneurones in the visceral ganglion is also indicated on the drawing of the larvae. At the neural plate stage, only the neural plate is shown and the dark blue ovals represent the secondary muscle lineage.