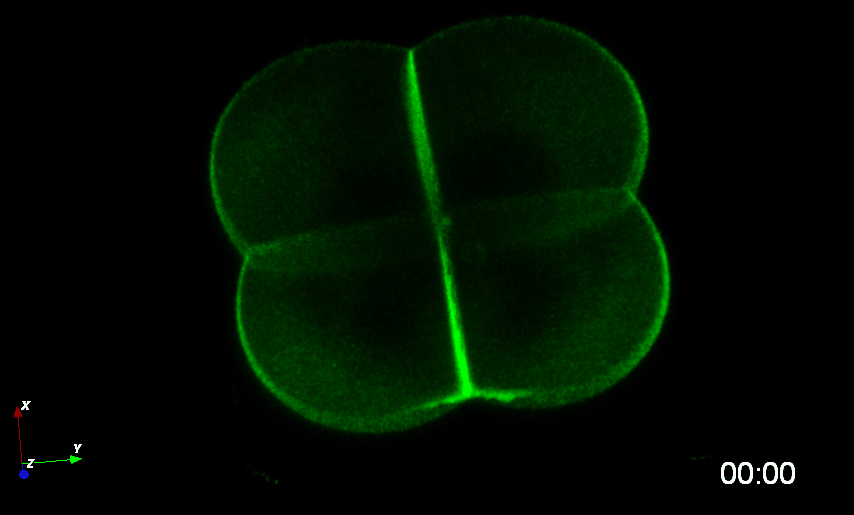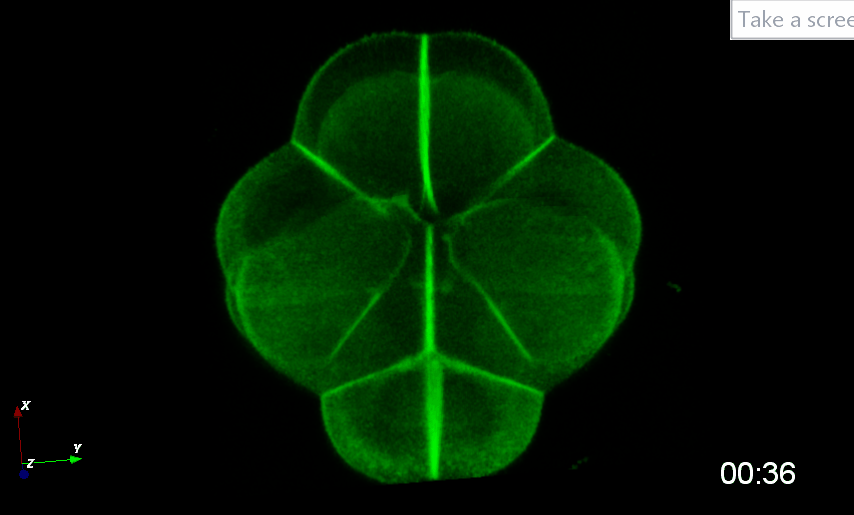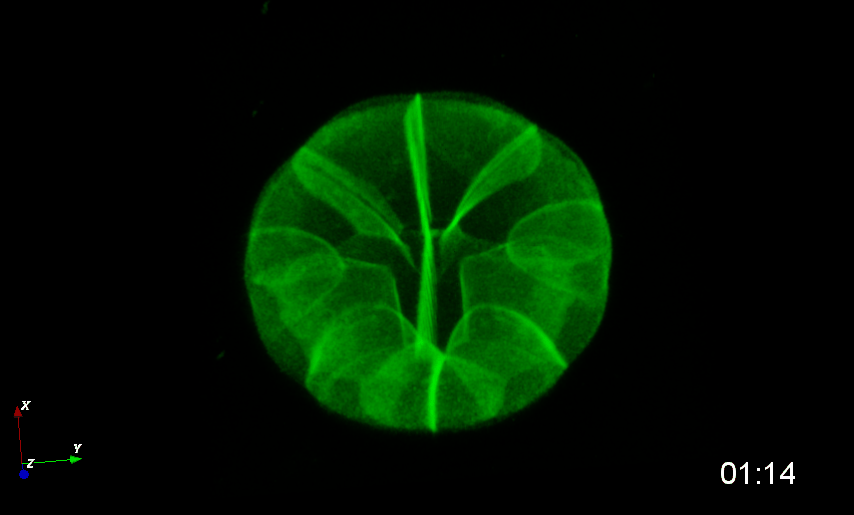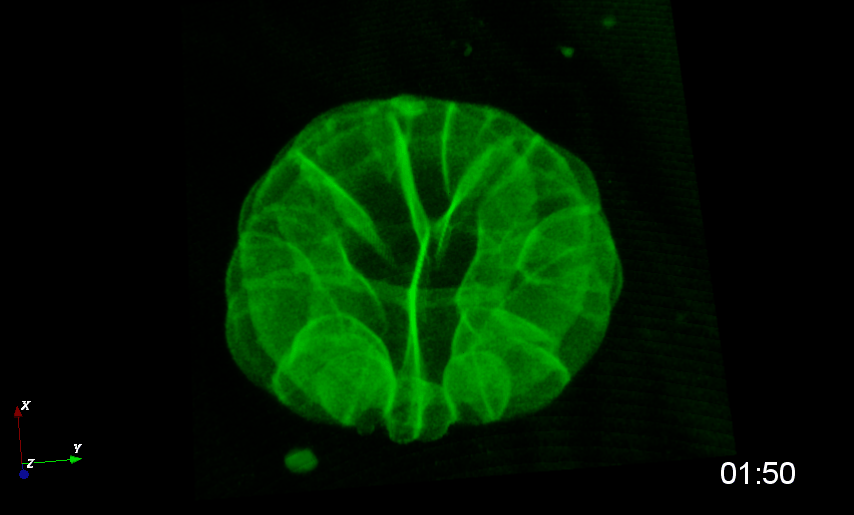
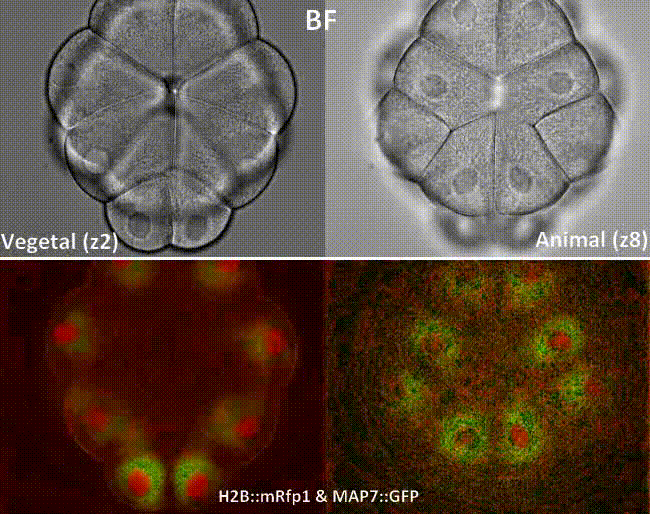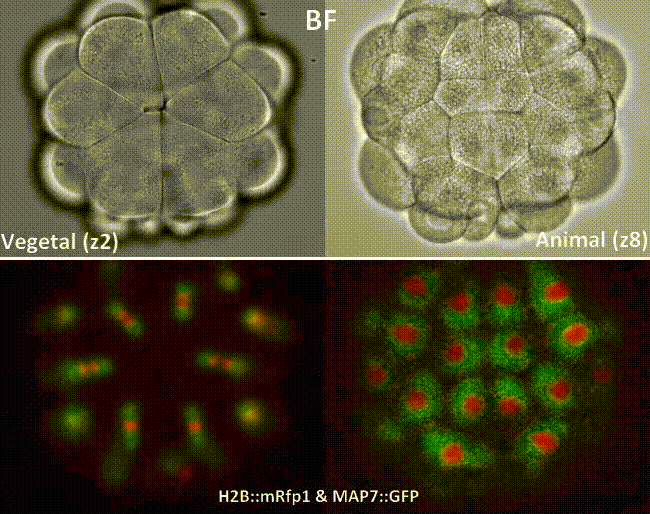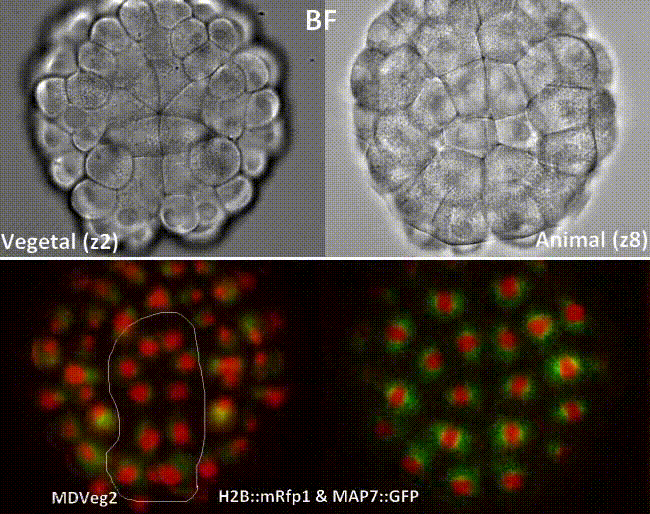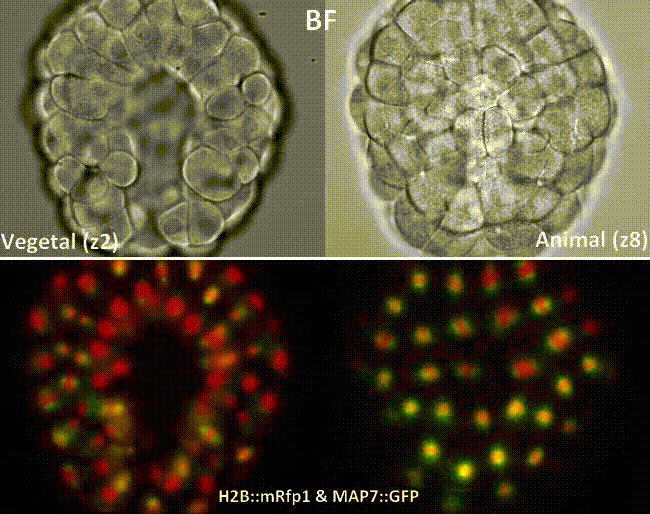
 Dumollard et al., 2013
Dumollard et al., 2013
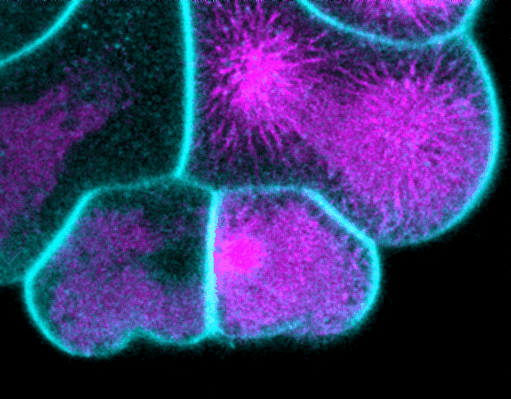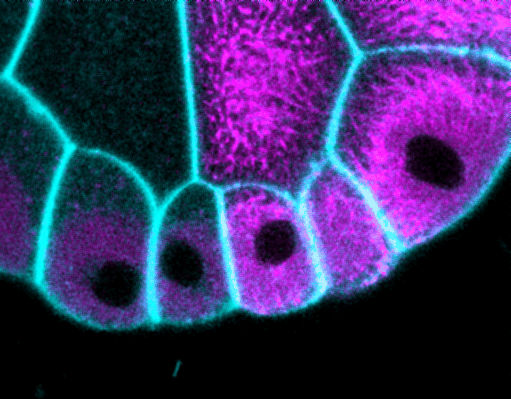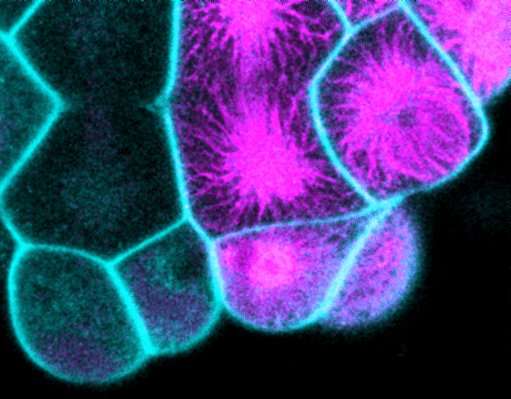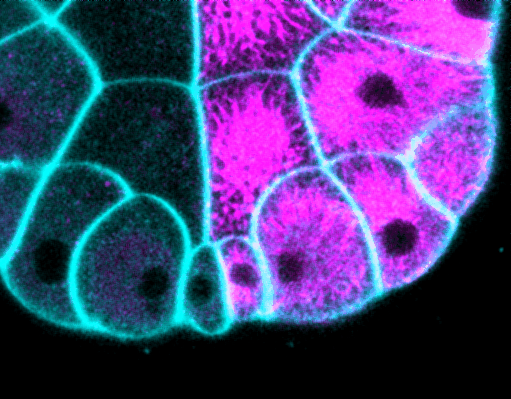 Costache et al. 2017
Costache et al. 2017
Dumollard et al., 2017
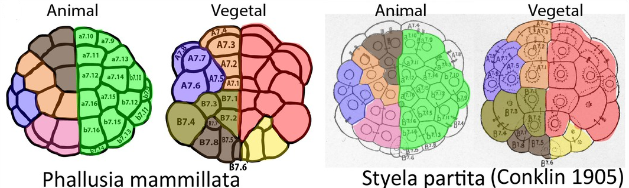
Cell position
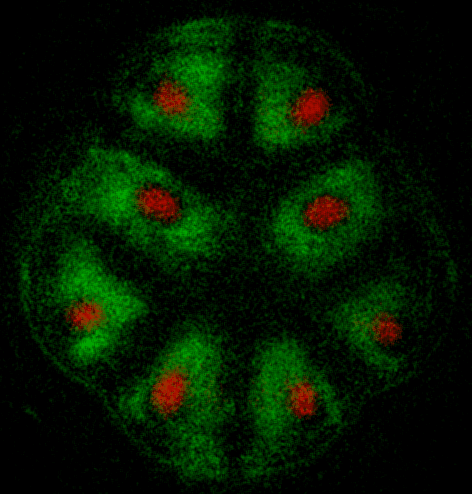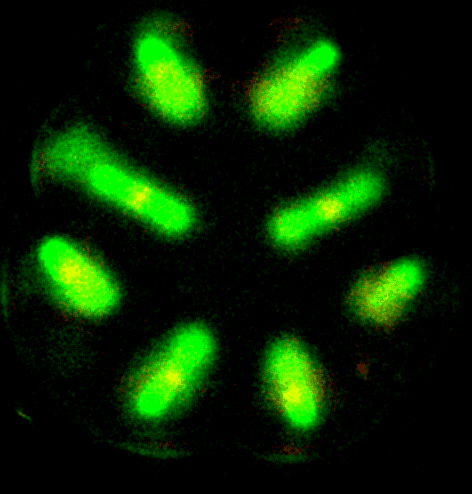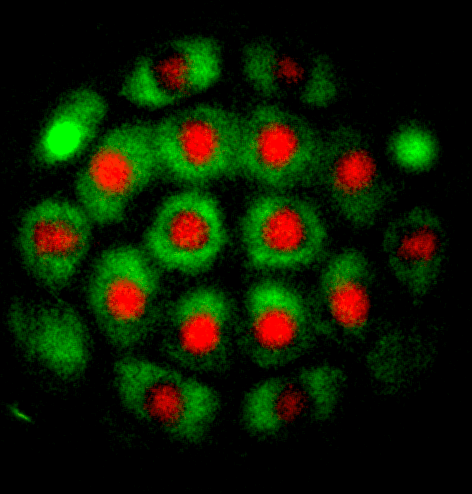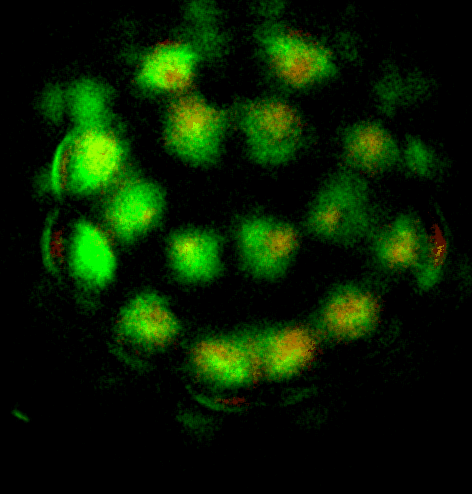 16-32 cell spindle rotation
16-32 cell spindle rotation

Laboratoire de Biologie du développement UMR 7009
Cette UE se déroule sur 2 semaines et a lieu au laboratoire de Biologie du Développement de Villefranche -sur-Mer (LBDV). Elle inclut l’examen de l’UE d’analyse scientifique (5V089) suivie par les étudiants de la spécialité de Biologie du Développement.
Durant la 1ère semaine, les étudiants participent à des ateliers et des rencontres avec les chercheurs du laboratoire.
Durant la 2ème semaine, les étudiants sont répartis dans les équipes pour y réaliser un mini projet qu’ils présentent le dernier jour du cours. Le cours est donné en anglais pour tout ou partie.
Modalités d’évaluation
Présentation orale du mini-projet (binôme, 100 %)
UE en anglais (partiellement ou totalement)
Master BMC, S3, 6 ECTS
Code UE: 5V200
Responsable de l’UE: Carine BARREAU (MCU): carine.barreau [at] obs-vlfr.fr
Cette UE permet aux étudiants de 1ère année de Master de passer 2 semaines à l’Observatoire Océanologique de Villefranche-sur-Mer. Le cours est obligatoire pour les étudiants du Master Biologie Intégrative, parcours Biologie et Bioressources Marines (BBMA) tandis qu’il peut être choisi en option par les étudiants du Master Biologie Moléculaire et Cellulaire (BMC). Les étudiants participent à des ateliers de présentation des organismes marins utilisés par les équipes de recherche du laboratoire (LBDV) et apprennent à les manipuler au cours de travaux pratiques dont les thématiques vont de la Biologie du Développement fondamentale à la toxicologie appliquée.
Modalités d’évaluation
Analyse d’article et présentation orale (50%)
Compte-rendu écrit de TP (50%)
UE partiellement en anglais
Master BIP, S2, 6 ECTS
Code UE: 4B022 (ouverte au Master BMC)
Responsable de l’UE: Carine BARREAU (MCU): carine.barreau [at] obs-vlfr.fr
Cette UE complémentaire se déroule sur 2 semaines et permet aux étudiants de découvrir les différents aspects (métiers & recherche) de l’Observatoire Océanologique de Villefranche-sur-Mer (OOV). Ateliers et journal clubs sont organisés afin que les étudiants mettent en pratique leurs connaissances théoriques en biologie et développent leur capacité de communication scientifique en français et en anglais.